7. フリーウェア¶
運用終了
TSUBAME3 は既に運用を終了しています。 TSUBAME4 のマニュアル類はこちら
フリーウェアの一覧表を以下に示します。
| ソフトウェア名 | 概要 |
|---|---|
| GAMESS | ソルバ・シミュレータ |
| Tinker | ソルバ・シミュレータ |
| GROMACS | ソルバ・シミュレータ |
| LAMMPS | ソルバ・シミュレータ |
| NAMMD | ソルバ・シミュレータ |
| QUANTUM ESPRESSO | ソルバ・シミュレータ |
| CP2K | ソルバ・シミュレータ |
| OpenFOAM | ソルバ・シミュレータ、可視化 |
| CuDNN | GPUライブラリ |
| NCCL | GPUライブラリ |
| Caffe | DeepLearningフレームワーク |
| Chainer | DeepLearningフレームワーク |
| TensorFlow | DeepLearningフレームワーク |
| DeePMD-kit | MD用DeepLearningフレームワーク |
| R | インタプリタ(Rmpi,rpudに対応) |
| clang | コンパイラ |
| Apache Hadoop | 分散データ処理ツール |
| POV-Ray | 可視化 |
| ParaView | 可視化 |
| VisIt | 可視化 |
| turbovnc | リモートGUI(X11) 表示 |
| gnuplot | データ可視化 |
| Tgif | 画像表示・編集 |
| GIMP | 画像表示・編集 |
| ImageMagick | 画像表示・編集 |
| TeX Live | TeX ディストリビューション |
| Java SDK | 開発環境 |
| PETSc | リニアシステムソルバ、ライブラリ |
| FFTW | 高速フーリエ変換ライブラリ |
| DMTCP | チェックポイント・リスタート |
| Singularity | Linux container for HPC |
7.1. 量子化学/MD関連ソフトウェア¶
7.1.1. GAMESS¶
GAMESSはオープンソースの第一原理分子量子化学計算アプリケーションです。
バッチキューシステムを利用したGAMESSの利用方法の例を以下に示します。
#!/bin/bash
#$ -cwd
#$ -l f_node=1
#$ -l h_rt=0:10:0
#$ -N gamess
. /etc/profile.d/modules.sh
module load intel intel-mpi gamess
cat $PE_HOSTFILE | awk '{print $1}' > $TMPDIR/machines
cd $GAMESS_DIR
./rungms exam08 mpi 4 4
詳細な説明は以下に記載されています。
http://www.msg.ameslab.gov/gamess/index.html
7.1.2. Tinker¶
Tinkerはバイオポリマーの為の特別な機能を備えた、分子動力学の為のモデリングソフトウェアです。
バッチキューシステムを利用したTinkerの利用方法の例を以下に示します。
#!/bin/bash
#$ -cwd
#$ -l f_node=1
#$ -l h_rt=0:10:0
#$ -N tinker
. /etc/profile.d/modules.sh
module load intel tinker
cp -rp $TINKER_DIR/example $TMPDIR
cd $TMPDIR/example
dynamic waterbox.xyz -k waterbox.key 100 1 1 2 300
cp -rp $TMPDIR/example $HOME
詳細な説明は以下に記載されています。
https://dasher.wustl.edu/tinker/
7.1.3. GROMACS¶
GROMACSは分子動力学シミュレーションとエネルギー最小化を行う為のエンジンです。
バッチキューシステムを利用したGROMACSの利用方法の例を以下に示します。
#!/bin/bash
#$ -cwd
#$ -l f_node=1
#$ -l h_rt=0:10:0
#$ -N gromacs
. /etc/profile.d/modules.sh
module load cuda/11.2.146 intel-mpi python/3.11.2 gcc/10.2.0 gromacs
cp -rp $GROMACS_DIR/examples/water_GMX50_bare.tar.gz $TMPDIR
cd $TMPDIR
tar xf water_GMX50_bare.tar.gz
cd water-cut1.0_GMX50_bare/3072
gmx_mpi grompp -f pme.mdp
OMP_NUM_THREADS=2 mpiexec.hydra -np 4 gmx_mpi mdrun
cp -rp $TMPDIR/water-cut1.0_GMX50_bare $HOME
詳細な説明は以下に記載されています。
7.1.4. LAMMPS¶
LAMMPSは液状、固体状、気体状の粒子の集団をモデル化する古典分子動力学コードです。
バッチキューシステムを利用したLAMMPSの利用方法の例を以下に記します。
#!/bin/bash
#$ -cwd
#$ -l f_node=1
#$ -l h_rt=0:10:0
#$ -N lammps
. /etc/profile.d/modules.sh
module load intel cuda openmpi/3.1.4-opa10.10-t3 ffmpeg python/3.11.2 lammps
cp -rp $LAMMPS_DIR/examples/VISCOSITY $TMPDIR
cd $TMPDIR/VISCOSITY
mpirun -x PATH -x LD_LIBRARY_PATH -x PSM2_CUDA=1 -np 4 lmp -pk gpu 0 -in in.gk.2d
cp -rp $TMPDIR/VISCOSITY $HOME
詳細な説明は以下に記載されています。
7.1.5. NAMD¶
NAMDは、大規模な生体分子システムの高性能シミュレーション用にデザインされたオブジェクト指向の並列分子動力学コードです。
バッチキューシステムを利用したNAMDの利用方法の例を以下に記します。
#!/bin/bash
#$ -cwd
#$ -l f_node=1
#$ -l h_rt=0:10:0
#$ -N namd
. /etc/profile.d/modules.sh
module load cuda intel namd
cp -rp $NAMD_DIR/examples/stmv.tar.gz $TMPDIR
cd $TMPDIR
tar xf stmv.tar.gz
cd stmv
namd3 +idlepoll +p4 +devices 0,1,2,3 stmv.namd
cp -rp $TMPDIR/stmv $HOME
Info
バージョン2以前ではコマンド名がnamd2となっておりますので、古いバージョンを使う場合にはnamd3をnamd2に置き換えて下さい。
詳細な説明は以下に記載されています。
https://www.ks.uiuc.edu/Research/namd/3.0/ug/
7.1.6. CP2K¶
CP2Kは固体、液体、分子、周期的、物質、結晶、生物系の原子シミュレーションを実行できる量子化学、固体物理ソフトウェアパッケージです。
バッチキューシステムを利用したCP2Kの利用方法の例を以下に記します。
#!/bin/bash
#$ -cwd
#$ -l f_node=1
#$ -l h_rt=0:10:0
#$ -N cp2k
. /etc/profile.d/modules.sh
module load cuda gcc openmpi/3.1.4-opa10.10-t3 cp2k
cp -rp $CP2K_DIR/benchmarks/QS $TMPDIR
cd $TMPDIR/QS
export OMP_NUM_THREADS=1
mpirun -x PATH -x LD_LIBRARY_PATH -x PSM2_CUDA=1 -np 4 cp2k.psmp -i H2O-32.inp -o H2O-32.out
cp -rp $TMPDIR/QS $HOME
詳細な説明は、以下に記載されています。
7.1.7. QUANTUM ESPRESSO¶
QUANTUM ESPRESSOは第一原理電子構造計算と材料モデリングのためのスイートです。
バッチキューシステムを利用したQUANTUM ESPRESSOの利用方法の例を以下に記します。
#!/bin/sh
#$ -cwd
#$ -l h_rt=00:10:00
#$ -l f_node=1
#$ -N q-e
. /etc/profile.d/modules.sh
module purge
module load cuda/10.2.89 pgi openmpi/3.1.4-opa10.10-t3 quantumespresso
cp -p $QUANTUMESPRESSO_DIR/test-suite/pw_scf/scf.in .
cp -p $QUANTUMESPRESSO_DIR/example/Si.pz-vbc.UPF .
mpirun -x ESPRESSO_PSEUDO=$PWD -x PATH -x LD_LIBRARY_PATH -x PSM2_CUDA=1 -x PSM2_GPUDIRECT=1 -np 4 pw.x < scf.in
詳細な説明は、以下に記載されています。
https://www.quantum-espresso.org/
7.2. CFD関連ソフトウェア¶
7.2.1. OpenFOAM¶
OpenFOAMはオープンソースの流体/連続体シミュレーションコードです。
Foudation版(openfoam)とESI版(openfoam-esi)の2種類がインストールされています。
バッチキューシステムを利用したOpenFOAMの利用方法の例を以下に記します。
#!/bin/bash
#$ -cwd
#$ -l f_node=1
#$ -l h_rt=0:10:0
#$ -N openform
. /etc/profile.d/modules.sh
module load cuda openmpi openfoam
mkdir -p $TMPDIR/$FOAM_RUN
cd $TMPDIR/$FOAM_RUN
cp -rp $FOAM_TUTORIALS .
cd tutorials/incompressible/icoFoam/cavity/cavity
blockMesh
icoFoam
paraFoam
ESI版OpenFOAMをご利用の場合は上記のmodule loadの箇所をmodule load cuda openmpi openfoam-esiとして下さい。
詳細な説明は以下に記載されています。
https://openfoam.org/resources/
http://www.openfoam.com/documentation/
7.3. GPU用数値計算ライブラリ¶
7.3.1. cuBLAS¶
cuBLASはGPUで動作するBLAS(Basic Linear Algebra Subprograms)ライブラリです。
利用方法
$ module load cuda
$ nvcc -gencode arch=compute_60,code=sm_60 -o sample sample.cu -lcublas
通常のC言語のプログラム中で、cuBLASを呼び出す場合、コンパイル時に-I、-L、-lで指定する必要があります。
$ module load cuda
$ gcc -o blas blas.c -I${CUDA_HOME}/include -L${CUDA_HOME}/lib64 -lcublas
7.3.2. cuSPARSE¶
cuSPARSEはNVIDIA GPU上で疎行列計算を行うためのライブラリです。
利用方法
$ module load cuda
$ nvcc -gencode arch=compute_60,code=sm_60 sample.cu -lcusparse -o sample
通常のC言語のプログラム中で、cuSPARSEを呼び出す場合、コンパイル時に-I、-L、-lで指定する必要があります。
$ module load cuda
$ g++ sample.c -lcusparse_static -I${CUDA_HOME}/include -L${CUDA_HOME}/lib64 -lculibos -lcudart_static -lpthread -ldl -o sample
7.3.3. cuFFT¶
cuFFTはNVIDIA GPU上で並列FFT(高速フーリエ変換)を行うためのライブラリです。
利用方法
$ module load cuda
$ nvcc -gencode arch=compute_60,code=sm_60 -o sample sample.cu -lcufft
通常のC言語のプログラム中で、cuFFTを呼び出す場合、コンパイル時に-I、-L、-lで指定する必要があります。
$ module load cuda
$ gcc -o blas blas.c -I${CUDA_HOME}/include -L${CUDA_HOME}/lib64 -lcufft
7.4. 機械学習、ビックデータ解析関連ソフトウェア¶
7.4.1. CuDNN¶
CuDNNはGPUを用いたDeep Neural Networkの為のライブラリです。
CuDNNの利用方法を以下に記します。
$ module load cuda cudnn
7.4.2. NCCL¶
NCCLは複数GPUの為の集団通信ライブラリです。
NCCLの利用方法の例を以下に記します。
$ module load cuda nccl
7.4.3. Caffe¶
CaffeはオープンソースのDeepLearningフレームワークです。
Caffeの利用方法の例を以下に記します。
$ module load cuda nccl cudnn/6.0 intel caffe/1.0
詳細な説明は以下に記載されています。
http://caffe.berkeleyvision.org/
MKLを利用する際はコードの前段に#define USE_MKLを追記し、$MKLROOT以下にある計算ライブラリを呼び出してください。
7.4.4. Chainer¶
Chainerはフレキシブルなニューラルネットワークのフレームワークです。
Chainerの利用方法の例を以下に記します。
$ module load intel cuda nccl/2.2.13 cudnn/7.1 openmpi/2.1.2 chainer/4.3.0
詳細な説明は以下に記載されています。
https://docs.chainer.org/en/stable/
7.4.5. TensorFlow¶
TensorFlowはデータフローグラフを用いた機械学習・AIのオープンソースのライブラリです。
TensorFlowの利用方法の例を以下に記します。
Python2.7の場合
$ module load python-extension
Python3.9.2の場合
$ module load python/3.9.2 cuda/11.2.146 cudnn/8.1 nccl/2.8.4 tensorflow
詳細な説明は以下に記載されています。
7.4.6. DeePMD-kit¶
DeePMD-kitはMD用機械学習フレームワークです。
DeePMD-kitのジョブスクリプトの例を以下に記します。
7.4.6.1 DeePMD-kit + LAMMPS¶
7.4.6.1.1. DeePMD-kit LAMMPS 1ノード¶
DeePMD-kit + LAMMPSのジョブスクリプト例(1ノード、4GPU)を以下に示します。
#!/bin/sh
#$ -l h_rt=6:00:00
#$ -l f_node=1
#$ -cwd
. /etc/profile.d/modules.sh
module purge
module load deepmd-kit/2.1.5 intel ffmpeg lammps/23jun2022_u2
module li 2>&1
# enable DeePMD-kit for lammps/23jun2022_u2
export LAMMPS_PLUGIN_PATH=$DEEPMD_KIT_DIR/lib/deepmd_lmp
# https://tutorials.deepmodeling.com/en/latest/Tutorials/DeePMD-kit/learnDoc/Handson-Tutorial%28v2.0.3%29.html
wget https://dp-public.oss-cn-beijing.aliyuncs.com/community/CH4.tar
tar xf CH4.tar
cd CH4/00.data
python3 <<EOF
import dpdata
import numpy as np
data = dpdata.LabeledSystem('OUTCAR', fmt = 'vasp/outcar')
print('# the data contains %d frames' % len(data))
# random choose 40 index for validation_data
index_validation = np.random.choice(200,size=40,replace=False)
# other indexes are training_data
index_training = list(set(range(200))-set(index_validation))
data_training = data.sub_system(index_training)
data_validation = data.sub_system(index_validation)
# all training data put into directory:"training_data"
data_training.to_deepmd_npy('training_data')
# all validation data put into directory:"validation_data"
data_validation.to_deepmd_npy('validation_data')
print('# the training data contains %d frames' % len(data_training))
print('# the validation data contains %d frames' % len(data_validation))
EOF
export PSM2_DEVICES="shm,self,hfi"
cd ../01.train
dp train input.json
dp freeze -o graph.pb
dp compress -i graph.pb -o graph-compress.pb
dp test -m graph-compress.pb -s ../00.data/validation_data -n 40 -d results
cd ../02.lmp
ln -s ../01.train/graph-compress.pb
lmp -i in.lammps
7.4.6.1.2. DeePMD-kit LAMMPS 2ノード¶
DeePMD-kit + LAMMPSのジョブスクリプト例(2ノード、8GPU)を以下に示します。
#!/bin/sh
#$ -l h_rt=12:00:00
#$ -l f_node=2
#$ -cwd
. /etc/profile.d/modules.sh
module purge
module load deepmd-kit/2.1.5 intel ffmpeg lammps/23jun2022_u2
module li 2>&1
# enable DeePMD-kit
export LAMMPS_PLUGIN_PATH=$DEEPMD_KIT_DIR/lib/deepmd_lmp
# https://tutorials.deepmodeling.com/en/latest/Tutorials/DeePMD-kit/learnDoc/Handson-Tutorial%28v2.0.3%29.html
wget https://dp-public.oss-cn-beijing.aliyuncs.com/community/CH4.tar
tar xf CH4.tar
cd CH4/00.data
python3 <<EOF
import dpdata
import numpy as np
data = dpdata.LabeledSystem('OUTCAR', fmt = 'vasp/outcar')
print('# the data contains %d frames' % len(data))
# random choose 40 index for validation_data
index_validation = np.random.choice(200,size=40,replace=False)
# other indexes are training_data
index_training = list(set(range(200))-set(index_validation))
data_training = data.sub_system(index_training)
data_validation = data.sub_system(index_validation)
# all training data put into directory:"training_data"
data_training.to_deepmd_npy('training_data')
# all validation data put into directory:"validation_data"
data_validation.to_deepmd_npy('validation_data')
print('# the training data contains %d frames' % len(data_training))
print('# the validation data contains %d frames' % len(data_validation))
EOF
export PSM2_DEVICES="shm,self,hfi"
cd ../01.train
mpirun -x PATH -x LD_LIBRARY_PATH -x PYTHONPATH -x PSM2_CUDA=1 -x NCCL_BUFFSIZE=1048576 -npernode 4 -np 8 dp train input.json
dp freeze -o graph.pb
dp compress -i graph.pb -o graph-compress.pb
dp test -m graph-compress.pb -s ../00.data/validation_data -n 40 -d results
cd ../02.lmp
ln -s ../01.train/graph-compress.pb
mpirun -x PATH -x LD_LIBRARY_PATH -x PYTHONPATH -x LAMMPS_PLUGIN_PATH -x PSM2_CUDA=1 -npernode 4 -np 8 lmp -i in.lammps
7.4.6.2 DeePMD-kit + GROMACS¶
DeePMD-kit + GROMACSのジョブスクリプト例(1ノード、4GPU)を以下に記します。
#!/bin/sh
#$ -l h_rt=8:00:00
#$ -l f_node=1
#$ -cwd
. /etc/profile.d/modules.sh
module purge
module load deepmd-kit/2.1.5 gromacs-deepmd/2020.2
module li 2>&1
export PSM2_DEVICES="shm,self,hfi"
cp -pr $DEEPMD_KIT_DIR/examples/examples/water .
cd water/se_e2_a
dp train input.json
dp freeze -o graph.pb
dp compress -i graph.pb -o graph-compress.pb
dp test -m graph-compress.pb -s ../data/data_3 -n 40 -d results
cd ../gmx
ln -s ../se_e2_a/graph-compress.pb frozen_model.pb
export GMX_DEEPMD_INPUT_JSON=input.json
gmx_mpi grompp -f md.mdp -c water.gro -p water.top -o md.tpr -maxwarn 3
gmx_mpi mdrun -deffnm md
gmx_mpi rdf -f md.trr -s md.tpr -o md_rdf.xvg -ref "name OW" -sel "name OW"
詳細な説明は以下に記載されています。
https://docs.deepmodeling.com/projects/deepmd/en/master/index.html
7.4.7. R¶
Rはデータ解析とグラフィックスの為のインタプリタ型プログラミング言語です。
並列処理用にRmpi、GPU用にrpudがインストールされています。
Rの利用方法の例を以下に記します。
$ module load intel cuda openmpi r
$ mpirun -stdin all -np 2 R --slave --vanilla < test.R
7.4.8. clang¶
clangはLLVMバックエンドのC/C++コンパイラです。
clangでGPU offloadするバイナリを生成する例を以下に示します。
- Cの場合
$ module load cuda clang
$ clang -fopenmp -fopenmp-targets=nvptx64-nvidia-cuda --cuda-path=$CUDA_HOME -Xopenmp-target -march=sm_60 test.c
- C++の場合
$ module load cuda clang
$ clang++ -stdlib=libc++ -fopenmp -fopenmp-targets=nvptx64-nvidia-cuda --cuda-path=$CUDA_HOME -Xopenmp-target -march=sm_60 test.cxx -lc++abi
詳細な説明は以下に記載されています。
7.4.9. Apache Hadoop¶
Apache Hadoopソフトウェアライブラリは単純なプログラミングモデルを用いて大きなデータセットを分散処理する為のフレームワークです。
Apache Hadoopの利用方法の例を以下に記します。
$ module load jdk hadoop
$ mkdir input
$ cp -p $HADOOP_HOME/etc/hadoop/*.xml input
$ hadoop jar $HADOOP_HOME/share/hadoop/mapreduce/hadoop-mapreduce-examples-2.8.0.jar grep input output 'dfs[a-z.]+'
$ cat output/part-r-00000
1 dfsadmin
バッチキューシステムの場合の利用手順を以下に示します。
#!/bin/bash
#$ -cwd
#$ -l f_node=1
#$ -l h_rt=0:10:0
#$ -N hadoop
. /etc/profile.d/modules.sh
module load jdk hadoop
cd $TMPDIR
mkdir input
cp -p $HADOOP_HOME/etc/hadoop/*.xml input
hadoop jar $HADOOP_HOME/share/hadoop/mapreduce/hadoop-mapreduce-examples-2.8.0.jar grep input output 'dfs[a-z.]+'
cp -rp output $HOME
7.5. 可視化関連ソフトウェア¶
7.5.1. POV-Ray¶
POV-Rayはフリーの光線追跡ソフトです。
POV-Rayの利用方法の例を以下に記します。
$ module load pov-ray
$ povray -benchmark
詳細な説明は以下に記載されています。
7.5.2. ParaView¶
ParaViewはオープンソース、マルチプラットフォームのデータ解析と可視化アプリケーションです。
ParaViewの利用方法の例をを以下に記します。
$ module load cuda openmpi paraview
$ paraview
7.5.2.1. 複数GPUを用いて可視化する場合¶
paraview/5.10.0、paraview/5.10.0-egl、を用いて複数ノードで複数GPUを用いて可視化することができます。
paraview/5.10.0-eglにはparaviewコマンドが含まれていないことにご注意ください。
以下はf_node=2で8GPUを使う例です。
- wrap.sh
#!/bin/sh
num_gpus_per_node=4
mod=$((OMPI_COMM_WORLD_RANK%num_gpus_per_node))
if [ $mod -eq 0 ];then
export VTK_DEFAULT_EGL_DEVICE_INDEX=0
elif [ $mod -eq 1 ];then
export VTK_DEFAULT_EGL_DEVICE_INDEX=1
elif [ $mod -eq 2 ];then
export VTK_DEFAULT_EGL_DEVICE_INDEX=2
elif [ $mod -eq 3 ];then
export VTK_DEFAULT_EGL_DEVICE_INDEX=3
fi
$*
- job.sh
#!/bin/sh
#$ -cwd
#$ -V
#$ -l h_rt=8:0:0
#$ -l f_node=2
. /etc/profile.d/modules.sh
module purge
module load cuda openmpi/3.1.4-opa10.10-t3 paraview/5.10.0-egl
mpirun -x PSM2_CUDA=1 -x PATH -x LD_LIBRARY_PATH -npernode 4 -np 8 ./wrap.sh pvserver
openmpi/3.1.4-opa10.10-t3モジュールを使って-x PSM2_CUDA=1としないとGPUで可視化部分だけが実行されGPGPU部分は実行されないのでご注意ください。
wrap.shに実行権限を忘れずに付与して下さい。(chmod 755 wrap.sh)
上記のジョブスクリプトで
qsub -g <グループ名> job.sh
を行い、ジョブを投入します。
qstatでジョブが流れているのを確認します。
yyyyyyyy@login0:~> qstat
job-ID prior name user state submit/start at queue jclass slots ja-task-ID
------------------------------------------------------------------------------------------------------------------------------------------------
xxxxxxx 0.55354 job.sh yyyyyyyy r 05/31/2020 09:24:19 all.q@rXiYnZ 56
ジョブが流れているノードにX転送でsshし、paraviewを起動します。
yyyyyyyy@login0:~> ssh -CY rXiYnZ
yyyyyyyy@rXiYnZ:~> module load cuda openmpi paraview/5.10.0
paraview
※turbovncを用いても可能です。
起動後、「File」->「Connect」をクリックし、「Add Server」をクリックします。
「Name」を適当に入力し(ここでは"test"とします)、「Configure」をクリックします。
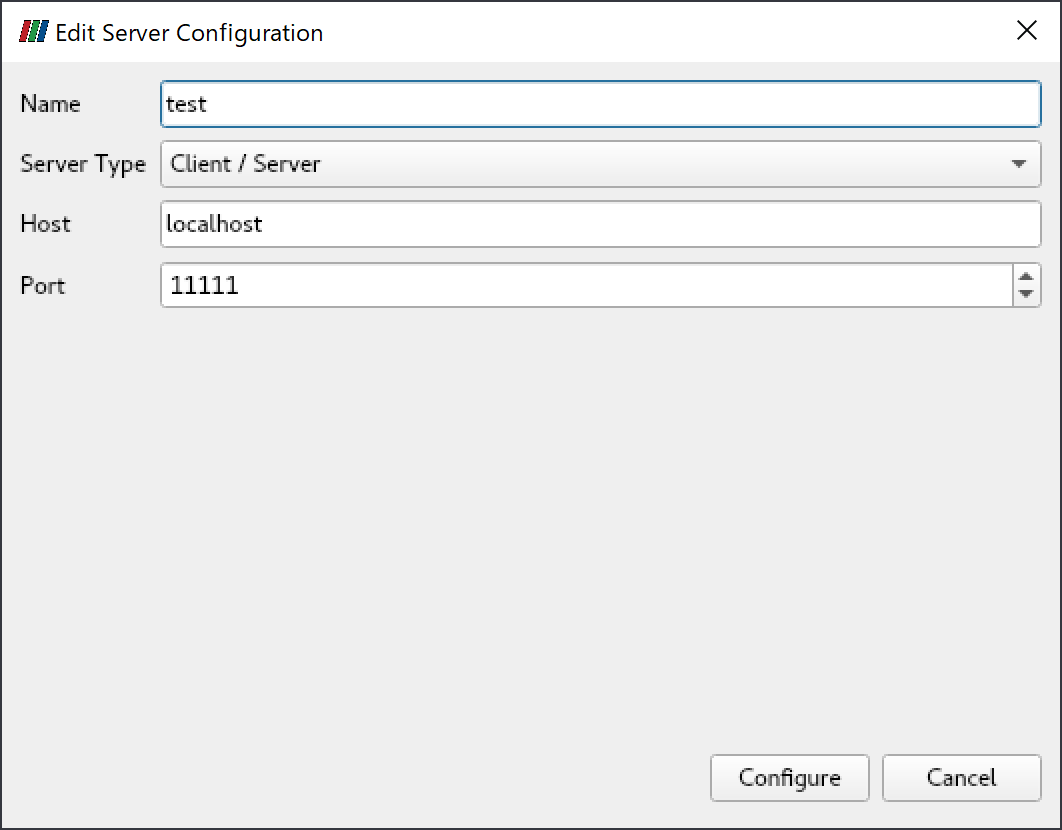
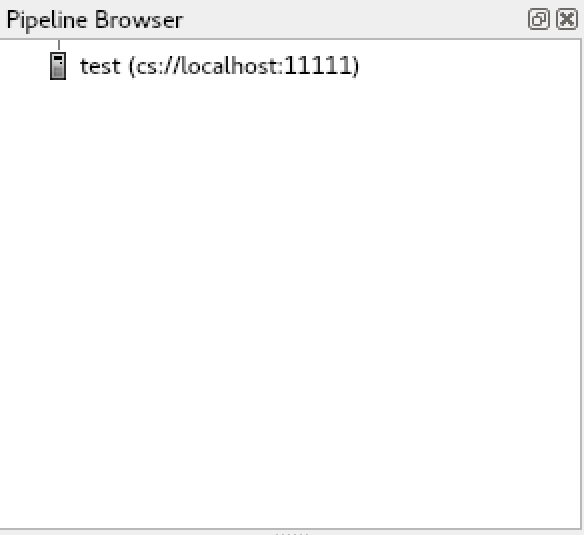
その後、「Connect」をクリックします。
接続されると、「Pipeline Browser」の項目にtest(cs://localhost:11111)が表示されます。
paraviewのサンプルデータはここからダウンロードすることができます。
詳細な説明は以下に記載されています。
7.5.3. VisIt¶
VisItはオープンソースの可視化アプリケーションです。
VisItの利用方法の例を以下に記します。
$ module load cuda openmpi vtk visit
$ visit
詳細な説明は以下に記載されています。
https://wci.llnl.gov/simulation/computer-codes/visit/
7.6. その他フリーウェア¶
7.6.1. turbovnc¶
turbovncはオープンソースのVNCソフトウェアです。
turbovncの使用方法の例を以下に記します。
※qrshで計算ノードを確保し計算ノード上で実行して下さい。
- 計算ノードを確保する
$ qrsh -g <グループ名> -l <資源タイプ>=<個数> -l h_rt=<時間>
- 確保した計算ノード上で以下を実行し、vncserverを起動する
$ module load turbovnc
$ vncserver
You will require a password to access your desktops.
Password: # <-- パスワードを聞かれるので設定する
Verify:
Would you like to enter a view-only password (y/n)? n
Desktop 'TurboVNC: rXiYnZ:1 ()' started on display rXiYnZ:1 # <-- ここのVNCのディスプレイ番号:1を覚えておく
Creating default startup script /home/n/xxxx/.vnc/xstartup.turbovnc
Starting applications specified in /home/n/xxxx/.vnc/xstartup.turbovnc
Log file is /home/n/xxxx/.vnc/rXiYnZ:1.log
画面サイズを大きくしたい場合はvncserver -geometry <WIDTH>x<HEIGHT>としてサイズを指定します。
- その後https://sourceforge.net/projects/turbovnc/files/から自分のPC用のインストーラをダウンロードし、turbovnc viewerをインストールする
- 計算ノードに接続したターミナルソフトからSSHポート転送の設定でローカルのポート5901を計算ノードのポート5901にポート転送するように設定する(もしディスプレイ番号がrXiYnZ:nだった場合、ポート転送のポート番号は5900+nに設定する)
- 自分のPCからturbovnc viewerを起動し、localhost:5901に接続して設定したパスワードを入力する
7.6.1.1. MobaXtermからVNCクライアントを利用する方法¶
MobaXtermにはVNCクライアントが内蔵されておりますので、VNCクライアントをインストールしなくてもVNC接続がご利用できます。
- qrshでノードを確保後、MobaXtermから「Sessions」->「New session」->「VNC」を選択する。
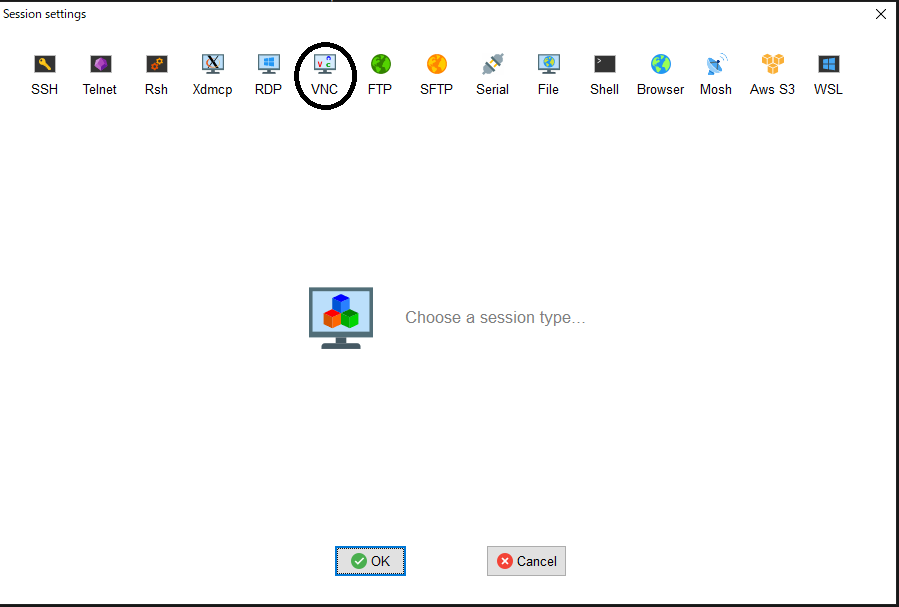
- その後、「Basic Vnc settings」の「Remote hostname or IP address」に確保した計算ノードのホスト名、「Port」を5900+nを入力し、「Network settings」の「Connect through SSH gateway(jump host)」をクリックし「Gateway SSH server」にlogin.t3.gsic.titech.ac.jpを入力、「Port」は22のまま、「User」に自分のTSUBAMEのログイン名を入力、「Use private key」にチェックを入れ自分の秘密鍵を入力する。
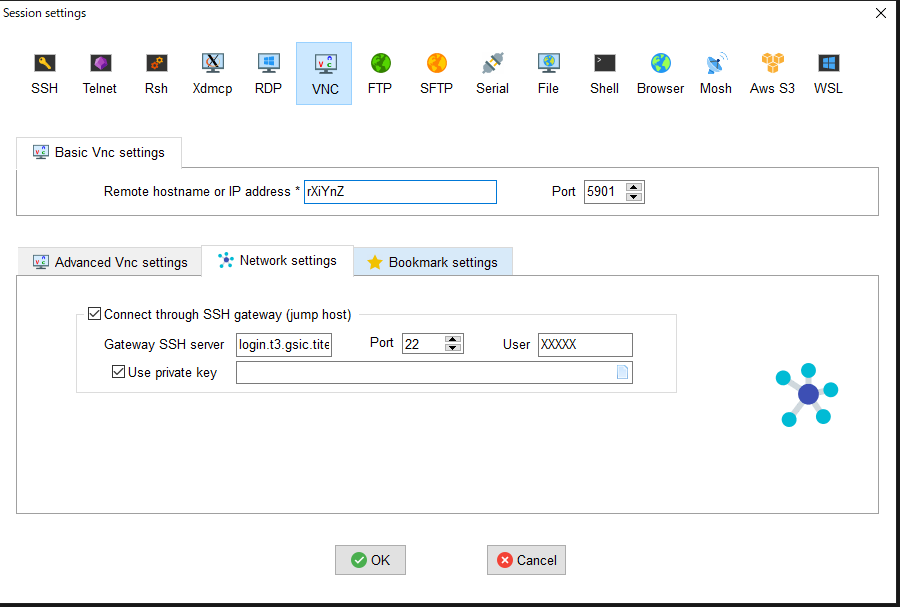
OKをクリックするとVNCクライアントが起動します。
7.6.1.2. turbovnc + VirtualGL¶
turbovnc用時に、GPUを1つ以上確保する資源タイプ(s_gpu, q_node, h_node, f_node)を用いている場合、VirtualGLを使用してGPUを用いて可視化することができます。
一部のアプリケーションではX転送でも通常のVNCでも描画に失敗するものがあります(例えばGpuTestやUNIGINE等)が、そのようなアプリケーションを実行したい場合はVirtualGLをお試し下さい。
例として、s_gpuの場合のVirtualGLの使用例を以下に示します。
$ qrsh ... -l s_gpu=1
$ . /etc/profile.d/modules.sh
$ module load turbovnc
$ Xorg -config xorg_vgl.conf :99 & # :99はVirtualGL用の任意のディスプレイ番号
$ vncserver
Warning
VirtualGLに使うディスプレイ番号はVNCで割り当てられたディスプレイ番号とは異なるので注意してください。
もし既に選択したディスプレイ番号が他のユーザに使われてしまっている場合、Xorgの実行で以下のようなエラーになります。
user@r7i7n7:~> Xorg -config xorg_vgl.conf :99 &
user@r7i7n7:~> (EE)
Fatal server error:
(EE) Server is already active for display 99
If this server is no longer running, remove /tmp/.X99-lock
and start again.
(EE)
(EE)
Please consult the The X.Org Foundation support
at http://wiki.x.org
for help.
(EE)
この場合は、:99を:100にするなどして他のユーザに使われていないディスプレイ番号を割り当てる用にしてください。
- VNCクライアントで接続し、以下を実行
$ vglrun -d :99 <OpenGLアプリケーション> # :99は上のXorgで設定したVirtualGL用のディスプレイ番号
複数GPUを確保していて、2番目以降のGPUを使用したい場合はディスプレイ番号にスクリーン番号を追加します。
$ vglrun -d :99.1 <OpenGLアプリケーション> # :99は上のXorgで設定したVirtualGL用のディスプレイ番号、 .1はスクリーン番号
上記でスクリーン番号を.2にするとGPU3番、.3にするとGPU4番が使われます。
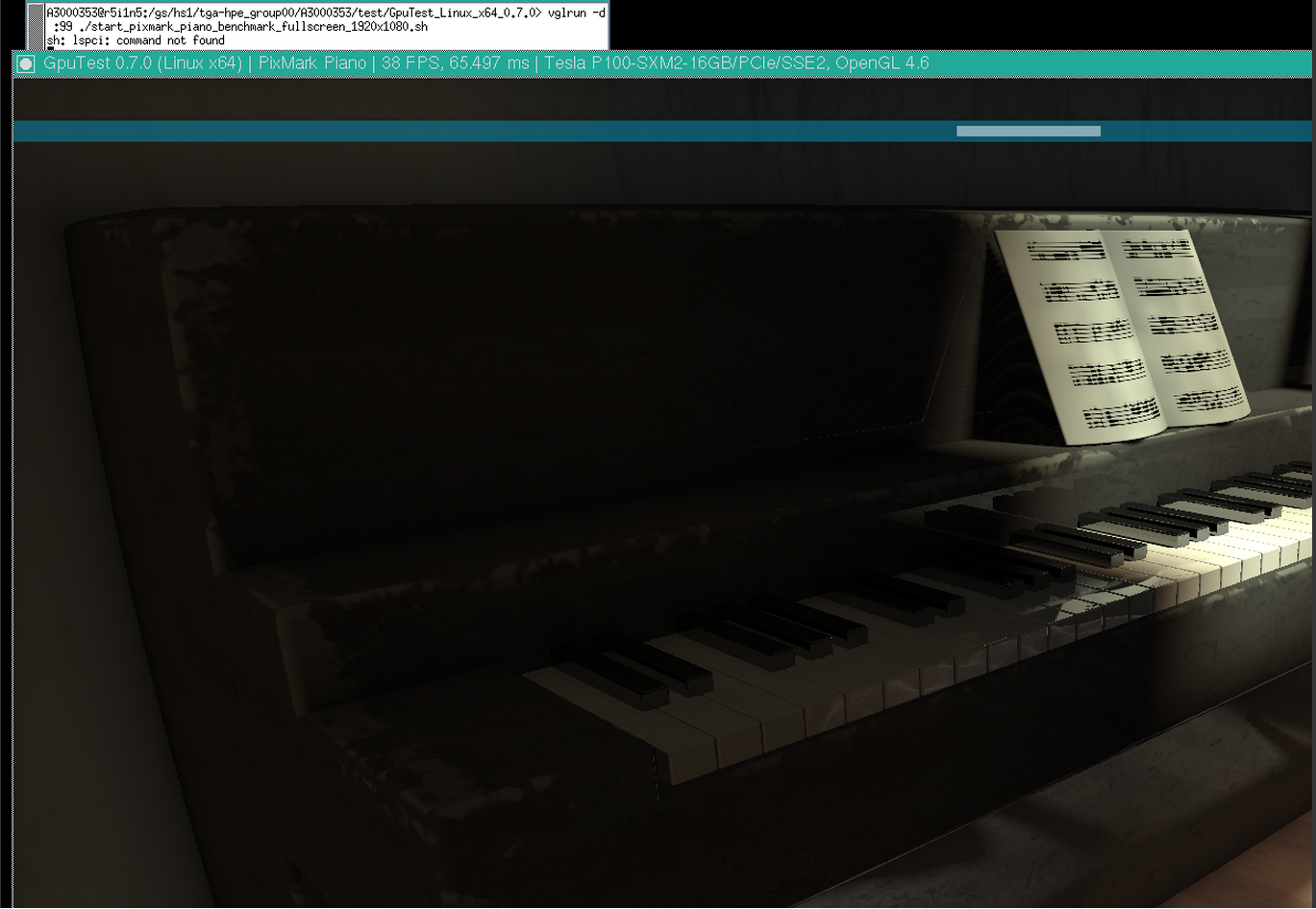
- GpuTestをVirtualGLを用いて実行する例
vglrun -d :99 ./start_pixmark_piano_benchmark_fullscreen_1920x1080.sh
7.6.2. gnuplot¶
gnuplotはコマンドラインのインタラクティブなグラフ描画プログラムです。
標準機能に加え、X11、latex、PDFlib-lite、Qt4に対応するようにビルドされています。
gnuplotの利用方法の例を以下に記します。
$ module load gnuplot
$ gnuplot
7.6.3. Tgif¶
tgifはオープンソースの描画ツールです。
tgifの利用方法を以下に記します。
$ module load tgif
$ tgif
※Cannot open the Default(Msg) Font '--courier-medium-r-normal--14-----*-iso8859-1'.というエラーが出て起動しない場合は、 ~/.Xdefaultsに以下の行を追加して下さい。
Tgif.DefFixedWidthFont: -*-fixed-medium-r-semicondensed--13-*-*-*-*-*-*-*
Tgif.DefFixedWidthRulerFont: -*-fixed-medium-r-semicondensed--13-*-*-*-*-*-*-*
Tgif.MenuFont: -*-fixed-medium-r-semicondensed--13-*-*-*-*-*-*-*
Tgif.BoldMsgFont: -*-fixed-medium-r-semicondensed--13-*-*-*-*-*-*-*
Tgif.MsgFont: -*-fixed-medium-r-semicondensed--13-*-*-*-*-*-*-*
7.6.4. GIMP¶
GIMPはオープンソースの画像操作プログラムです。
GIMPの利用方法の例を以下に記します。
$ module load gimp
$ gimp
7.6.5. ImageMagick¶
ImageMagickは画像処理ツールです。
標準機能に加え、X11、HDRI、libwmf、jpegに対応するようにビルドされています。
ImageMagickの利用方法の例を以下に記します。
$ module load imagemagick
$ convert -size 48x1024 -colorspace RGB 'gradient:#000000-#ffffff' -rotate 90 -gamma 0.5 -gamma 2.0 result.jpg
7.6.6. pLaTeX2e¶
pLaTeX2eは日本語化されたLaTex2eの一つです。
pLaTeX2eの利用方法の例を以下に記します。
$ module load texlive
$ platex test.tex
$ dvipdfmx test.dvi
※pdfの作成にはdvipdfmxをご利用ください。dvipdfでは日本語が正常に変換されません。
7.6.7. Java SDK¶
Java SDKとして、Oracle JDK 1.8がインストールされています。
Java SDKの利用方法の例を以下に記します。
$ module load jdk
$ javac Test.java
$ java Test
7.6.8. PETSc¶
PETScはオープンソースの並列数値計算ライブラリです。線型方程式の求解等を行うことができます。
実数用、複素数用の2種類がインストールされています。
PETScの利用方法の例を以下に記します。
$ module load intel intel-mpi
$ module load petsc/3.7.6/real ← 実数用
又は
$ module load petsc/3.7.6/complex ← 複素数用
$ mpiifort test.F -lpetsc
7.6.9. FFTW¶
FFTWはオープンソースの高速フーリエ変換用ライブラリです。
FFTW 2x系列と3x系列は非互換な為、バージョン2系と3系の2種類がインストールされております。
FFTWの利用方法の例を以下に記します。
$ module load intel intel-mpi fftw ← Intel MPIの場合
又は
$ module load intel cuda openmpi fftw ← Open MPIの場合
$ ifort test.f90 -lfftw3
7.6.10. DMTCP¶
DMTCPはマルチノード・マルチスレッド対応のチェックポイントツールです。
DMTCPの利用方法の例を以下に記します。
- チェックポイントを作成する場合
#!/bin/sh
# 他の指定については記載を省略
module load dmtcp
export DMTCP_CHECKPOINT_DIR=<イメージの保存先>
export DMTCP_COORD_HOST=`hostname`
export DMTCP_CHECKPOINT_INTERVAL=<チェックポイント取得間隔>
dmtcp_coordinator --quiet --exit-on-last --daemon 2>&1 # DMTCPの実行
dmtcp_launch ./a.out # DMTCP経由でプログラムの実行
- 作成されたチェックポイントからリスタートする場合
#!/bin/sh
# 他の指定については記載を省略
module load dmtcp
export DMTCP_CHECKPOINT_DIR=<イメージの保存先>
export DMTCP_COORD_HOST=`hostname`
export DMTCP_CHECKPOINT_INTERVAL=<チェックポイント取得間隔>
# DMTCP_CHECKPOINT_INTERVALの間隔でDMTCP_CHECKPOINT_DIRにチェックポイントが作成されます。
# dmtcp_restart_script.shスクリプトでチェックポイントからプログラムをリスタートできます。
$DMTCP_CHECKPOINT_DIR/dmtcp_restart_script.sh # イメージからのリスタート
DMTCPについては以下のページを参照ください。
http://dmtcp.sourceforge.net/
7.6.11. Singularity¶
SingularityはHPC向けLinuxコンテナです。
Singularityの使い方の例を以下に記します。
※qrshでノードを確保した後に実行して下さい。
シェルを起動する場合
$ module load singularity
$ cp -p $SINGULARITY_DIR/image_samples/centos/centos7.6-opa10.9.sif .
$ singularity shell --nv -B /gs -B /apps -B /scr centos7.6-opa10.9.sif
コンテナ内のコマンドを実行する場合
$ module load singularity
$ cp -p $SINGULARITY_DIR/image_samples/centos/centos7.6-opa10.9.sif .
$ singularity exec --nv -B /gs -B /apps -B /scr centos7.6-opa10.9.sif <コマンド>
MPIを実行する場合
$ module load singularity cuda openmpi
$ cp -p $SINGULARITY_DIR/image_samples/centos/centos7.6-opa10.9.sif .
$ mpirun -x LD_LIBRARY_PATH -x SINGULARITYENV_LD_LIBRARY_PATH=$LD_LIBRARY_PATH -x SINGULARITYENV_PATH=$PATH -x <環境変数> -npernode <プロセス数/ノード> -np <プロセス数> singularity exec --nv -B /apps -B /gs -B /scr/ centos7.6-opa10.9.sif <MPI実行バイナリ>
singularity/3.4.2以降では、fakerootオプションを使ってユーザ権限でコンテナを編集することができます。
Info
Singularityがfakerootオプションに対応したのは3.3.0ですが、TSUBAMEの環境に起因する問題があり、3.4.1までのSingularityでは同オプションが正常に動作しません。
fakerootオプションでイメージを編集するには計算ノードにて$T3TMPDIR上で実行する必要があります。
以下にcentosのイメージにvimをインストールする例を記します。
$ cd $T3TMPDIR
$ module load singularity
$ singularity build -s centos/ docker://centos:latest
INFO: Starting build...
Getting image source signatures
...
$ singularity shell -f -w centos # -f がfakerootオプションです
Singularity> id
uid=0(root) gid=0(root) groups=0(root)
Singularity> unset TMPDIR # "Cannot create temporary file - mkstemp: No such file or directory"のエラーの回避策です
Singularity> yum install -y vim
Failed to set locale, defaulting to C.UTF-8
CentOS-8 - AppStream 6.6 MB/s | 5.8 MB 00:00
CentOS-8 - Base 5.0 MB/s | 2.2 MB 00:00
CentOS-8 - Extras
...
Installed:
gpm-libs-1.20.7-15.el8.x86_64 vim-common-2:8.0.1763-13.el8.x86_64 vim-enhanced-2:8.0.1763-13.el8.x86_64
vim-filesystem-2:8.0.1763-13.el8.noarch which-2.21-12.el8.x86_64
Complete!
Singularity> which vim
/usr/bin/vim
Singularity> exit
$ singularity build -f centos.sif centos/
INFO: Starting build...
INFO: Creating SIF file...
INFO: Build complete: centos.sif
$ singularity shell centos.sif
Singularity> which vim
/usr/bin/vim # <--- vimがインストールされています
コンテナイメージにCUDA版OPAドライバライブラリをインストールする方法(centos7.5にCUDA版OPA10.9.0.1.2をインストールする場合)
※システムメンテナンスによってOPAが更新されることがございますので、新しいバージョンのOPAに更新された場合はは適時バージョンを変更して下さい。
OPAのバージョンは以下で確認が可能です。
$ rpm -qa |grep opaconfig
opaconfig-10.9.0.1-2.x86_64
このリンクから対応するOSのOPAのインストーラをダウンロードしてください
$ module load singularity/3.4.2
$ cp -p IntelOPA-IFS.RHEL75-x86_64.10.9.0.1.2.tgz ~
$ singularity build -s centos7.5/ docker://centos:centos7.5.1804
$ find centos7.5/usr/ -mindepth 1 -maxdepth 1 -perm 555 -print0 |xargs -0 chmod 755 # イメージ内の一部のファイル/ディレクトリに書き込み権限がないので追加します
$ singularity shell -f -w centos7.5
Singularity centos:~> tar xf IntelOPA-IFS.RHEL75-x86_64.10.9.0.1.2.tgz
Singularity centos:~> cd IntelOPA-IFS.RHEL75-x86_64.10.9.0.1.2/IntelOPA-OFA_DELTA.RHEL75-x86_64.10.9.0.1.2/RPMS/redhat-ES75/CUDA/
Singularity centos:~> yum install -y numactl-libs hwloc-libs libfabric libibverbs infinipath-psm
Singularity centos:~> rpm --force -ivh libpsm2-*.rpm
Singularity centos:~> exit
$ find centos7.5/usr/bin -perm 000 -print0 |xargs -0 chmod 755 # 上記yum install後、なぜかパーミッションが000のファイルが/usr/binにインストールされていますので変更します
$ singularity build centos7.5.sif centos7.5/
上記ではIFS版を用いていますが、BASIC版をダウンロードしても構いません。
詳細な説明は以下に記載されています。
apptainerを使用する場合はmodule load apptainerとし、コマンドのsingularityの部分をapptainerに変更してください。
7.6.12. Alphafold¶
Alphafoldは機械学習を用いたタンパク質構造予測プログラムです。
Alphafoldを利用する例を以下に示します。
-
初期設定 (ログインノードもしくは計算ノード)
module purge module load cuda/11.0.3 alphafold/2.0.0 cp -pr $ALPHAFOLD_DIR . cd alphafold git pull # 最新版に更新 # 特定のバージョンを使用したい場合(この場合はv2.0.1)は以下を追加してください。 # git checkout -b v2.0.1 v2.0.1 -
実行時 (alphafold/2.0.0のジョブスクリプトの例)
#!/bin/sh #$ -l h_rt=24:00:00 #$ -l f_node=1 #$ -cwd . /etc/profile.d/modules.sh module purge module load cuda/11.0.3 alphafold/2.0.0 module li cd alphafold ./run_alphafold.sh -a 0,1,2,3 -d $ALPHAFOLD_DATA_DIR -o dummy_test/ -m model_1 -f ./example/query.fasta -t 2020-05-14 -
実行時 (alphafold/2.1.1のジョブスクリプトの例)
#!/bin/sh #$ -l h_rt=24:00:00 #$ -l f_node=1 #$ -cwd . /etc/profile.d/modules.sh module purge module load cuda/11.0.3 alphafold/2.1.1 module li cd alphafold ./run_alphafold.sh -a 0,1,2,3 -d $ALPHAFOLD_DATA_DIR -o dummy_test/ -f ./example/query.fasta -t 2020-05-14
2.2.0の場合はalphafold/2.1.1のmodule load cuda/11.0.3 alphafold/2.1.1の部分をmodule load cuda/11.0.3 alphafold/2.2.0に変えて下さい。
データベースファイルの容量が大きいため、可能な限り個別にダウンロードすることはお避け下さい。
Alphafoldの詳細な説明は以下をご参照下さい。
https://github.com/deepmind/alphafold